Der Sinn der Gegensinn-RNA
Forschungsbericht (importiert) 2006 - Max-Planck-Institut für Infektionsbiologie
RNA wird wiederentdeckt
Die letzten Jahre haben deutlich gezeigt, dass RNA-Moleküle weit mehr biologische Funktionen haben als zuvor angenommen. Traditionell schrieb man nämlich der RNA nur drei grundlegende Rollen zu, nämlich i.) als Botenstoff der Erbinformation in Form von messenger RNAs (mRNAs), ii.) als genetisches Material bestimmter Viren und iii.) als essenzieller Bestandteil der Proteinbiosynthese-Maschinerie, der Ribosomen, dort in Form von transfer RNA (tRNA) und ribosomal RNA (rRNA). Dass RNA selbst wie Enzyme katalytisch aktiv sein kann und dass kleine, nicht-translatierte RNAs andere wichtige Funktionen in der Zelle, zum Beispiel beim Spleißen von mRNAs, haben, ist zwar schon länger bekannt, ebenso auch, dass nicht-kodierende RNA-Moleküle selbst als Regulatoren der Genexpression fungieren können. Dennoch schienen sich solche regulatorischen Funktionen hauptsächlich auf bakterielle Plasmide, Phagen oder Transposons zu beschränken. Das änderte sich jedoch vor etwa fünf Jahren, als eine systematische Suche in Bakterien und höheren Lebewesen einen ganzen Mikrokosmos an nicht-kodierenden, kleinen RNAs zutage förderte. Bald darauf gewann die Anwendung kleiner RNAs für das gezielte Ausschalten von Genen in höheren Organismen, die so genannte RNAi (RNA-Interferenz)-Technik enorm an Fahrt.
Waren im Modellbakterium Escherichia coli nur zehn kleine nichtkodierende RNAs (sRNAs) bekannt gewesen, stieg deren Zahl binnen kürzester Zeit auf nun über achtzig. Weitere hunderte sRNAs sind bisher vorausgesagt, müssen allerdings noch nachgewiesen werden. Die Funktion der meisten neu entdeckten sRNAs ist noch unbekannt, doch vieles weist darauf hin, dass sie eine neue Klasse von Regulatoren bei bakteriellen Stressantworten bilden.
sRNAs in krankheitserregenden Bakterien
Bisher sind nur wenige sRNAs in pathogenen Bakterien beschrieben worden. Im Gegensatz zur systematischen Suche im nicht-pathogenen E. coli-Stamm K12 wurden sie zumeist nur durch Zufall entdeckt und über ihre Funktion ist wenig bekannt. Die erfolgreiche Suche in E. coli beruhte größtenteils auf der Entdeckung, dass die sRNA-kodierenden Gene dieses Bakteriums in konservierter Form auch in eng verwandten pathogenen Enterobakterien, wie etwa Salmonella-, Klebsiella- und Yersinia-Spezies, vorhanden sind.
Im Mittelpunkt der Arbeitsgruppe RNA-Biologie steht die funktionelle Charakterisierung von sRNAs im Hinblick auf bakterielle Virulenz und die Wechselwirkung pathogener Bakterien mit befallenen Wirtszellen. Dabei wird ein ganzes Spektrum molekularer, biochemischer, genetischer und bioinformatischer Methoden angewendet. Da bisher nur in einigen wenigen Fällen tiefer gehend untersucht wurde, wie solche kleinen RNAs regulatorisch in die bakterielle Genexpression eingreifen, ist die Arbeitsgruppe besonders an den molekularen Mechanismen dieser Regulation interessiert.
Die meisten sRNAs operieren als so genannte antisense RNAs, das heißt sie können über Basenkomplementarität an Boten-RNAs binden und so die Synthese bestimmter Proteine steuern. Die Paarungen zwischen diesen Molekülen sind allerdings in ihrer Länge recht kurz, imperfekt und daher schwer vorherzusagen, womit die größte Herausforderung der Forschung darin besteht, den sRNAs ein oder mehrere Zielmoleküle zuzuordenen.
Kleine RNAs kontrollieren den Zustand der Zellhülle von Salmonellen
Zusammen mit britischen Wissenschaftlern wurde kürzlich der Mechanismus beschrieben, wie in Salmonella-Bakterien nicht-kodierende RNA-Moleküle den Zustand der Zellhülle überwachen [1]. Salmonellen sind Mensch und Tier befallende Bakterien, und als Krankheitserreger müssen sie sich fortlaufend und schnell neuen Umweltbedingungen anpassen können, um innerhalb und außerhalb ihrer Wirte zu überleben. Solche Anpassungen verursacht Stress, gegen den sich Salmonellen beispielsweise mit ihrer Zellhülle schützen. Die Zellhülle intakt zu halten und immer wieder neu zu ordnen ist äußerst wichtig für das Überleben der Salmonellen unter den widrigsten Bedingungen. Befallen zum Beispiel Salmonellen einen Wirt, müssen sie dem niedrigen pH-Wert des Magens genauso trotzen können wie der sauerstoffarmen und salzreichen Umgebung im Darm. Um sich anpassen zu können, müssen Salmonellen in der Lage sein, immer wieder neue und andere Membranproteine in ihre Zellhülle einzubauen. Diese äußeren Membranproteine bilden Poren und erlauben es den Bakterien, Nährstoffe und Salze aus ihrer Umgebung aufzunehmen - ein zweischneidiges Schwert, denn gleichzeitig werden diese Poren-Proteine auch vom Immunsystem des Wirts erkannt, ihre Synthese muss also durch das Bakterium genau kontrolliert werden, damit nicht zu viele davon in der Hülle akkumulieren.
Es wurde jetzt gefunden, dass Salmonellen in einer Art Überwachungsschleife ganz genau registrieren können, ob sich als Zeichen eines Schadens an ihrer Hülle missgefaltete Membranproteine anhäufen. Sobald dies registriert wird, schalten kleine nichtkodierende sRNAs sofort die Biosynthese von Membranproteinen ab (Abb. 1). Da im Gegensatz zu regulatorischen Proteinen die Synthese solcher sRNAs in wenigen Sekunden abgeschlossen ist, kann diese Schutzreaktion mit einer enorm hohen Geschwindigkeit ablaufen.
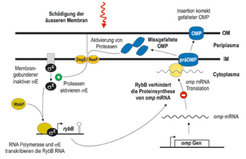
Die regulatorischen sRNAs lösen gleich noch ein zweites Problem, nämlich das der äußerst stabilen, Membranprotein kodierenden Boten-RNAs. Diese RNAs haben nämlich biologische Halbwertszeiten von etwa einer Viertelstunde - im Vergleich zu anderen Boten-RNAs mit Halbwertszeiten von wenigen Minuten. Was auch immer der Grund für diese große Stabilität sein mag, sie bedeutet, dass selbst wenn das entsprechende Gen schnell abgeschaltet wird, die Synthese des entsprechenden Membranproteins von der Boten-RNA noch lange weiter läuft – zu lange für Salmonellen, die, wie gesagt, oft innerhalb von Sekunden ihre Hülle neu ordnen müssen. Die regulatorischen sRNAs können dieses Problem lösen, indem sie direkt die Membranprotein kodierenden Boten-RNAs inaktivieren und so die Produktion von Hüllproteinen ganz abschalten.
Eine Besonderheit der neu entdeckten, regulatorischen sRNAs besteht darin, dass sie hochspezifisch die Membranprotein kodierenden Boten-RNAs erkennen können, obgleich diese selbst untereinander sehr verschieden sind. Wie genau diese Erkennung funktioniert, wird in den kommenden Jahren herauszufinden sein.
